Olá pessoal, hoje eu trago mais alguns apontamentos em biologia molecular, dessa vez sobre a abordagem proteômica e também sobre a biologia sintética.
Abordagem proteômica
Por que estudar proteínas?
- Para estudar a expressão de um gene: DNA -> RNA -> Proteína (produto final do gene);
- Alvo terapêutico;
- Desenvolvimento de novos fármacos;
- Vacinas;
- Aumentar a produtividade na agricultura.
Proteoma: conjunto de proteínas produzidas por uma célula em determinadas condições fisiológicas.
Proteômica: estuda e identifica esse conjunto de proteínas e relaciona com suas funções. É um passo seguinte à genômica, auxiliando os cientistas a compreenderem melhor questões sobre o mecanismo de certas doenças e as substâncias que poderão ser utilizadas para sua prevenção e combate.
Exemplo: o ovo, o pintinho e o galo => têm o mesmo genoma, mas as proteínas são diferentes em cada uma dessas fases do desenvolvimento da ave. Os genes que determinam a produção de penas ou da crista do galo são ativados em momentos diferentes e ''ordenam'' a produção de proteínas diferentes.
Exemplo: uma célula cancerosa possui um padrão metabólico diferente da célula típica. Uma expressão de genes diferentes da célula típica. O estudo dessas diferenças pode revelar os genes envolvidos, o que é uma indicação precisa dos alvos metabólicos a serem estudados.
A complexidade da proteômica reside em descobrir as proteínas de um organismo e como elas funcionam em cada momento. Isso representa um potencial de cura de doenças, desenvolvimento de novos medicamentos ou aumento de produtividade de culturas agrícolas. O conhecimento da estrutura das proteínas permite compreender sua função nas células.
Para analisar a expressão gênica, podem ser utilizadas proteínas extraídas de diferentes tipos de tecido, como sangue, saliva, plasma, tecido sólido, tecidos em diferentes fases do desenvolvimento.
Metodologia:
- Separação de proteínas em eletroforese em géis de poliacrilamida bidimensionais, permite a análise individual das proteínas bem como sua quantificação diferencial;
- Sequenciamento proteico é possível através da espectrometria de massa, o que permite conhecimento maior sobre a sequência dos genes.
Métodos de purificação e caracterização de proteínas:
Cromatografia (chroma = cor; graphein = escrever):
A mistura de substâncias para o fracionamento é dissolvida em um líquido (fase móvel), que passa através de uma coluna contendo uma matriz sólida e porosa (fase estacionária).
As diferentes proteínas são retardas diferencialmente pela sua interação com a matriz e podem ser coletadas separadamente à medida que são eluídas na parte inferior da coluna.
Na cromatografia de troca iônica, a matriz carrega cargas iônicas que retardam o movimento das moléculas de carga oposta. DEAE-celulose (+), CM-celulose (-) e fosfocelulose (-).
Na cromatografia de filtração em gel, a matriz é porosa, moléculas pequenas penetram nos poros e sua migração é retardada enquanto moléculas grandes migram livremente.
- Requer pequenos volumes da amostra;
- Permite identificar diferentes compostos na mesma medição analítica;
- No final obtém-se um cromatograma com as seguintes características: cada composto deixa a coluna como um pico simétrico; cada composto aparece no cromatograma num tempo característico (tempo de retenção).
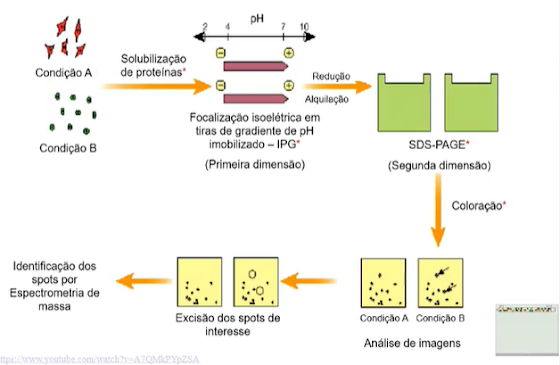
Proteômica: com o advento da genômica, a proteômica evidencia-se como mais uma técnica para análise de proteínas. Seus passos são:
- Solubilização de proteínas;
- Eletroforese bidimensional (2D);
- Análise de espectrometria de massa;
- Identificação de proteínas por métodos de bioinformática.
Solubilização de proteínas:
- ReadyPrep Sequential Extraction Kit (Bio-Rad);
- Pré-fracionamento de proteínas presentes em amostras -> solubilização diferencial.
Quantificação das proteínas:
- Método Colorimétrico de Bradford.
Eletroforese bidimensional (2D):
Método mais eficiente de separação simultânea de milhares de proteínas diferentes - análise de amostras complexas. Duas corridas eletroforéticas: a primeira dimensão é a focalização isoelétrica (IEF) baseada no ponto isoelétrico; a segunda dimensão é o SDS-PAGE baseado na massa molecular. Em seguida, ocorre a coloração do gel (Coomassie Blue, nitrato de prata etc.), e se produz mapas 2D (digitalizados) para análise da imagem com softwares especializados.
Em resumo:
- Focalização isoelétrica: primeira dimensão;
- SDS-PAGE: segunda dimensão;
- Coloração;
- Análise da imagem.
Eletroforese bidimensional (2D): primeira dimensão
A focalização isolelétrica separa as proteínas de acordo com seu ponto isoelétrico (pI).
A primeira dimensão consiste na focalização isoelétrica, método eletroforético que separa as proteínas de acordo com seu ponto isoelétrico. As proteínas são moléculas anfotéricas podendo apresentar carga líquida positiva, negativa ou neutra (= 0) dependendo do pH ao seu redor. O ponto isoelétrico de uma proteína é o pH onde a carga líquida da proteína é igual a 0.
Sobre a influência de um campo elétrico as proteínas migram em direção aos polos + ou -. Durante a migração, as proteínas vão se tornando menos carregadas até atingirem seu pI onde ficam imobilizadas.
Uma vez separadas por pI, as proteínas (na tira de poliacrilamida) são submetidas à SDS-PAGE, onde são separadas por massa molecular/peso molecular (PM).
- Equilíbrio da tira de IPG;
- Separação das proteínas na segunda dimensão: géis SDS-PAGE (10-15%) -> cuba de eletroforese vertical.
Uma molécula com carga elétrica desloca-se num campo magnético numa velocidade proporcional à sua densidade de carga total, tamanho e forma.
O termo SDS-PAGE refere-se à eletroforese em gel de poliacrilamida na presença de dodecil sulfato de sódio (SDS),em inglês: sodium dodecyl sulfate–polyacrylamide gel electrophoresis. As características desse sistema eletroforético são:
- Gel desnaturante;
- Rompe as pontes de hidrogênio e bloqueia as interações hidrofóbicas;
- Elimina as estruturas terciária e secundária das proteínas;
- Proteínas lineares.
 |
| Dentro dos círculos vermelhos estão exemplos de spots. |
Espectrometria de massa:
Os spots proteicos diferencialmente expressos são excisados do gel e passados para a espectrometria de massa.
Atualmente, existem dois métodos principais de ionização utilizados em proteômica, o MALDI (Matrix-Assisted Laser Desorption/Ionization) e o ESI (Electrospray Ionization), o primeiro empregado para amostras em estado sólido e o segundo para amostras em estado líquido (Figura 2). No método MALDI, os peptídeos são cocristalizados com uma matriz orgânica, geralmente ácido α-ciano-4- hidroxicinamínico. Após bombardeamento por laser, a matriz sublima e seus íons transferem a carga para os analitos, resultando na formação de íons peptídicos. Uma variante do MALDI denominada SELDI (Surface-Enhanced Laser Desorption/Ionization) é geralmente empregada para análise do proteoma de baixo peso molecular e utiliza várias matrizes ou chips que exploram as características cromatográficas e biofísicas das diferentes proteínas. Esses chips podem apresentar superfícies hidrofóbicas, de troca iônica ou com íons metálicos imobilizados, ou mesmo anticorpos, receptores, enzimas e ligantes com alta afinidade por proteínas específicas. Assim, após a lavagem dos compostos não ligados, uma matriz é colocada sobre o chip e os espectros são obtidos por ionização com laser. Outra variante do MALDI é o IMS (Imaging Mass Spectrometry), que permite a obtenção de dados de massas de peptídeos e proteínas diretamente de seções de tecidos biológicos. Esse método oferece importantes vantagens em relação à análise por imuno-histoquímica, incluindo rapidez e independência do uso de anticorpos.
No ESI, diferentemente do MALDI, uma solução aquosa com o analito é forçada a atravessar uma agulha capilar submetida à alta voltagem. A solução é ejetada como um aerossol de gotas altamente carregadas que, após evaporação do solvente por um fluxo de gás inerte aquecido, geram formas ionizadas do analito.
Independentemente do método de ionização, a massa molecular dos íons é avaliada em um analisador após passagem por uma câmara de vácuo. Os tipos mais comuns de analisadores são o TOF (Time Of Flight), o quadrupolo e o ion trap.
Nos analisadores TOF, os íons resultantes da primeira fase são acelerados por um potencial entre dois eletrodos e atravessam um tubo de vácuo com velocidade inversamente proporcional à sua massa. Quando os íons atingem o detector, o tempo decorrido entre a ionização e a detecção é utilizado para derivar o valor m/z. Na verdade, o detector converte o sinal da passagem do íon em sinal analógico, que é lido e interpretado por uma estação de trabalho. O resultado final é um gráfico de m/z versus intensidade (contagem de íons), comumente referido como espectro MS Os sinais gerados são comparados com informações disponíveis em bancos de dados como o MASCOT e o SEQUEST, o que permite identificar a proteína de interesse.
Uma das limitações do sistema MALDI-TOF é a dificuldade de detecção de proteínas de baixo peso molecular que geram, por causa dessa característica, poucos peptídeos. O sistema também não é capaz de detectar mais de um componente de uma mistura. Para melhorar o desempenho, os analisadores TOF podem ser combinados com analisadores quadrupolos (Qs), que apresentam um conjunto de quatro eletrodos em bastão e funcionam como filtros de massas. Entre esses eletrodos, um campo elétrico assegura que somente íons de uma determinada razão m/z sigam a trajetória ao detector enquanto os demais são desviados.
Os analisadores do tipo ion trap ou armadilha de íons (IT) filtram e aprisionam em um campo elétrico tridimensional íons de interesse, que são gradualmente liberados em ordem de m/z crescente. Os FT-ICRs (Fourier Transform Ion Cyclotron Resonance) são ion traps com um campo magnético adicional, que força os íons a exibirem um movimento circular com ciclos de alta frequência. O analisador determina a razão m/z a partir da frequência do movimento ciclotrônico utilizando a transformação de Fourier19. O orbitrap é outro tipo de analisador IT no qual os íons oscilam ao longo e ao redor de um eletrodo em forma de espiral. A frequência dessa oscilação é proporcional à raiz quadrada da razão massa/carga e pode ser determinada com alta precisão. Essa tecnologia migrou para sistemas híbridos de dois espectrômetros de massas independentes que reúnem, por exemplo, um ion trap e um orbitrap ou um ion trap e um FT-ICR.
Identificação de proteínas por métodos de bioinformática
- ''Mapa'' de peptídeos -> comparado com banco de dados de sequências não redundantes;
- Banco de dados: sequência de proteínas conhecidas -> simulação de digestão nas condições dos experimentos;
- Bancos de dados MASCOT e SEQUEST.
Análise proteômica em resumo:
Biologia sintética: uma área do século XXI
A biologia sintética é uma área que combina biologia, química e engenharia para projetar e construir novas funções e sistemas vivos, ou para redesenhar os sistemas vivos existentes com o propósito de torná-los mais úteis.
Bases tecnológicas:
- Tecnologia do DNA (síntese, sequenciamento e amplificação do DNA);
- Engenharia genética.
Como toda nova área do conhecimento, a biologia sintética exige uma avaliação dos riscos e das condutas a seguir, desde as três perspectivas hoje consideradas fundamentais:
- Biossegurança;
- Biosseguridade;
- Bioética.
Principais subáreas:
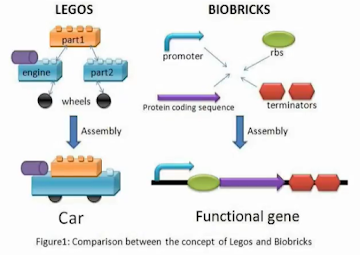
1. Construção de circuitos de DNA
- Partes: codificam funções básicas (sequência de uma proteína, promotor de RNA polimerase) e determinam a eficiência (PoPS e RiPS ou número de vezes/segundo que uma polimerase ou um ribossomo passam por determinado ponto);
- Dispositivos: coleções de partes que implementam uma função (produção de uma proteína fluorescente em resposta a uma substância presente no ambiente);
- Sistemas: tarefas complexas (oscilar entre duas cores com uma frequência determinada);
- Chassis: Escherichia coli, Saccharomyces cerevisiae etc.
- Democratização do conhecimento;
- Motivação das novas gerações para participar das atividades científicas;
- Estímulo da criatividade.
Incertezas:
- Sistemas construídos juntando partes e dispositivos seguros poderiam ser inseguros?
- Quais os riscos devido à participação de pessoas sem treinamento adequado (biohackers)?
- Quais os riscos devido à participação de pessoas mal intencionadas (biocrackers)?
- Poder-se-ia desenvolver uma bioeconomia ilícita?
- Quais os códigos de conduta e autorregulação das empresas que sintetizam DNA para evitar a disseminação de material que possa ser utilizado para elaborar armas biológicas ou toxinas? (Caso do poliovírus e do vírus da gripe espanhola).
2. Genomas mínimos e protocélulas:
Protocélulas: apesar de estarem formadas por dois componentes (RNA replicase e uma membrana de ácidos graxos), uma protocélula seria capaz de crescer, de se duplicar e de evoluir.
Quais seriam as primeiras aplicações?
- Área de saúde (Novartis e Synthetic Genomics Vaccines - CV): banco de DNA viral sintético que permita, em caso de epidemia, iniciar a produção de vacinas logo após a caracterização da cepa por World Health Organization; construção de uma vacina universal para o vírus da influenza, a partir de alguma sequência comum a todas as variantes;
- Área ambiental (Exxon Mobil e Craig Venter): biocumbustíveis a partir de algas.
Importância econômica:
- Cerca de 20% da indústria química poderia depender da biologia sintética (2015);
- Mercado da biologia sintética: US$ 10,8 bilhões (2016).
Certezas e incertezas:
- É a biologia sintética nada mais do que uma engenharia genética extrema? Se for assim, os riscos dos organismos gerados por biologia sintética (BS) e organismos geneticamente modificados (OGM) seriam análogos...
- Mas se não for assim...
- O que aconteceria com um organismo BS liberado no ambiente onde não existem predadores específicos?
- Poderia um microrganismo desenhado para cumprir uma função determinada de biorremediação ter efeito sobre outra substância presente no ambiente?
- Poderia haver intercâmbio genético entre um organismo sintético e um organismo biológico natural contaminando o pool gênico natural?
- Os organismos BS não poderiam ser avaliados como OGMs, mas como GSO (do inglês, Genetically Secured Organisms), e as medidas de contenção teriam que ser redefinidas: a ''certeza de contenção'' (do inglês, Certainty of Containment ou CoC), estabelece que a probabilidade de escapamento da contenção, e de disseminação e de interação não intencional com o ambiente deve ser virtualmente nula.
Como garantir a certeza de contenção (CoC)?
- Sistemas bioquímicos não naturais
- Alteração do tamanho do DNA com benzopurinas e benzopirimidinas (xDNA ou expanded DNA, YDNA ou wide DNA),
- Uso de ácidos xenonucleicos (XNAs),
- Extensão do alfabeto (ATCGPZ),
- Construção de ribossomos ortogonais para o reconhecimento de um código de 4 bases),
- Substituição de códons (TAG, substituído por TAA)
- Inclusão de aminoácidos não naturais.
• Qual seria a eficiência desses sistemas?
• Solucionariam o problema ou criariam outros novos?
A percepção pública:
• Preocupação central: as mudanças climáticas.
• A biologia sintética visa os setores de energia e medicamentos, setores pouco mobilizadores.
• As instituições reguladoras assumiram o estudo dos aspectos éticos, legais e sociais, anteriormente desenvolvidos pelos grupos opositores.
• Pouco interesse nas eventuais consequências socioeconômicas da biologia sintética.
• Pouco interesse nos aspectos relacionados com a propriedade intelectual.
• Algum interesse na ética relativa ao design de seres humanos.
FATORES NOVOS
• Sites de Internet e redes sociais promovendo a reflexão sobre o impacto da BS.
- THE SYNTHETIC BIOLOGY PROJECT (http://www.synbioproject.org/about/)
- SYNBIOSAFE (http://www.synbiosafe.eu/)
• Participação dos estudantes na promoção da Biologia Sintética.
O estímulo à criatividade nos laboratórios de garagem e a competições como IGEM por parte de instituições respeitadas como MIT, Harvard, Berkeley etc. criam um ambiente favorável. O livre acesso à informação e ao uso dos biobricks disponíveis e permitidos não tem volta atrás.
• Envolvimento de designers, artistas plásticos e cineastas.
Exposições e festivais de cinema temáticos, permitem a divulgação e a apresentação de diferentes formas de percepção da biologia sintética, favoráveis ou não.
- BIO:FICTION SCIENCE, FILMS AND ART FESTIVAL (http://bio-fiction.com/videos/?p=41)
- SUPER CELL ORGANIZATION/Super cell market (http://www.super-cell.org/)
É NECESSÁRIO REGULAR COM MAIS RIGOR?
• A "Presidential Commission for the Study of Bioethical Issues" recomenda um sistema regulatório incipiente de maneira a maximizar os benefícios e minimizar os riscos (EEUU, 2010). As recomendações ao presidente Barack Obama podem ser resumidas em uma vigilância prudente.
• Existe uma regulação para a Biologia Sintética (Estados Unidos e União Europeia)
• O consenso geral é de não haver necessidade de incrementar a regulação específica para a biologia sintética.
• Não é possível limitar o acesso a materiais e equipamentos. Tal como está crescendo a biologia sintética, em um sistema transparente de código aberto (open source), uma regulação estrita acabaria limitando o acesso ao saber
Mais de 100 organizações solicitam mais regulação e inclusive uma moratória.
SYNBIOWATCH
Considerações finais
Contudo, o desenvolvimento da Biologia Sintética deve ser acompanhado de perto, levando em conta três perspectivas:
- Biossegurança (combinações de biobricks, liberação no ambiente e sistemas de contenção, propriedade intelectual, sistemas ortogonais, medidas a tomar em caso de acidente etc.)
- Biosseguridade (responsabilidade das empresas que sintetizam DNA, possibilidade de mal uso de equipamentos e organismos etc.)
- Bioética (design de seres vivos, propriedade intelectual etc.)
Bibliografia
Referências online:
Separação de proteínas por eletroforese (SDS-PAGE). In: USP: e-disciplinas. Disponível em: https://edisciplinas.usp.br/pluginfile.php/5750299/mod_resource/content/1/Aula%204%20-%20SDS-PAGE.pdf. Acesso em: 24 nov. 2023.
Santos, R. M. Eletroforese em gel de poliacrilamdia (SDS-PAGE). In: Unesp - Câmpus de Jaboticabal Disponível em: https://www.fcav.unesp.br/Home/departamentos/patologia/HELIOJOSEMONTASSIER/sds-page-aula-imuno-vet.pdf. Acesso em: 24 nov. 2023.
Electrospray ionization(ESI): Simple Principle, working mechanism, 4 advantages. In: Chemist Notes. Disponível em: https://chemistnotes.com/analytical_chemistry/electrospray-ionizationesi/#google_vignette.Acesso em: 24 nov. 2023.
Matrix-Assisted Laser Desorption Ionization (MALDI). In: National High Magnetic Field Laboratory. Disponível em: https://nationalmaglab.org/user-facilities/icr/techniques/ionization-techniques/maldi/. Acesso em: 24 nov. 2023.
Barbosa, E. B., Vidotto, A., Polachini, G. M., Henrique, T., Marqui, A. B. T. de ., & Tajara, E. H.. (2012). Proteômica: metodologias e aplicações no estudo de doenças humanas. Revista Da Associação Médica Brasileira, 58(3), 366–375. Disponível em: https://doi.org/10.1590/S0104-42302012000300019. Acesso em: 24 nov. 2023.
Quecine, M. C. Genética Molecular e os microrganismos - Aula 9 - Genética Molecular. In: USP`- Departamento de Genética. Disponível em: https://edisciplinas.usp.br/pluginfile.php/5008494/mod_resource/content/2/Aula%209%20-%20GEN%C3%89TICA%20MOLECULAR%20APLICADA%20NO%20MELHORAMENTO%20DE%20MICRORGANISMOS%20E%20BIOLOGIA%20SINT%C3%89TICA%20%281%29.pdf.Acesso em: 24 nov. 2023.
































Nenhum comentário:
Postar um comentário