Olá pessoal, hoje eu trago mais alguns apontamentos em biologia molecular, essa publicação é uma continuação da publicação sobre sequenciamento e hibridização, aqui iremos tratar sobre metodologias para avaliação da expressão gênica (hibridizações, microarrays).
Metodologias para avaliação da expressão gênica
- Serial Analysis of Gene Expression (SAGE) (Velculescu et al., 1995);
- Diffential Display (DDRT-PCR) (Liang and Pardee, 1992);
- Macroarrays (Chen et al., 1998);
- Microarrays (Schena et al., 1995);
- Hibridização subtrativa (Jiang et al., 2001);
- PCR quantitativo ou em Tempo Real (Livak et al., 1995).
Chips de DNA ou microarranjos ou microarrays
É útil para investigar a expressão de genomas inteiros ou de grandes grupos de genes. Sequências específicas de nucleotídeos de genes ou sondas são aplicadas à membrana onde ocorrerá a hibridização, a hibridização ocorre com RNA ou cDNA radioativo ou fluorescente.
Vantagens:
- Milhares de genes podem ser comparados de uma só vez;
- As lâminas podem ser construídas usando genes completos ou fragmentos de genes;
- Análise de genes de células em diferentes condições, ex.: célula típica x célula cancerosa;
- Expressão de um mesmo gene em diferentes fases do desenvolvimento.
- cDNA controle e teste marcadas diferencialmente;
- Leitura feita por scanner;
- Bibliotecas de cDNA são hibridizadas juntas, em um só chip;
- Genes presentes em ambas as bibliotecas competirão pela ligação à sonda;
- Intensidade da fluorescência é diretamente proporcional à quantidade do gene presente em uma amostra comparada com a outra.
- Essas sequências representam frações de uma grande biblioteca de genes que estão presentes em uma célula;
- Se um gene estiver ativo dentro de uma célula, então o cDNA (produzido a partir do transcrito do mRNA) se ligará ao seu oligo complementar;
- Se o cDNA tiver sido marcado com fluorescência, então o oligo complementar pode ser identificado; Cada oligo representa uma sequência genética conhecida, portanto as doenças genéticas são identificadas de acordo com as quais os oligos são hibridizados;
- Quanto maior a expressão de um gene específico, mais brilhante será a fluorescência observada no poço de microarranjo relevante.
As novas e poderosas tecnologias de hibridização em microarranjo foram desenvolvidas nos anos 1990 para permitir a realização de ensaios paralelos em massa, nos quais milhares de ensaios de hibridização individuais são realizados de maneira simultânea e sob as mesmas condições experimentais. A hibridização em microarranjo foi primeiramente desenvolvida como uma ferramenta para mapeamento de larga escala e para o sequenciamento de DNA. Entretanto, a principal responsável pela evolução desta tecnologia foi a necessidade de analisar simultaneamente a expressão de um grande número de genes. À medida em que as bibliotecas de cDNA foram desenvolvidas e grandes números de clones de cDNA caracterizados se tornaram disponíveis, os pesquisadores começaram a planejar análises genômicas da expressão gênica. Ensaios de hibridização multiplex eram necessários para utilizar grandes números de sondas de DNA gene-específicas simultaneamente.
O princípio da técnica de microarranjo consiste na hibridização entre uma sonda (cDNA ou oligonucleotídeo) e uma molécula-alvo marcada com fluoróforo, usualmente um cDNA. As sondas contêm sequências complementares às moléculas-alvo correspondendo a uma parte específica de um gene e são fixadas em uma superfície sólida através de agulhas robotizadas, fotolitografia ou por impressão a jato. O RNA mensageiro extraído das amostras a serem analisadas são utilizados como moldes para a formação do cDNA. Este cDNA é então marcado com fluoróforo para posterior hibridização. Uma das aplicações da técnica de microarranjo mais utilizada é a comparação entre duas amostras distintas (por exemplo, uma amostra de doença e uma amostra saudável); nesta condição, moléculas de DNA provenientes das duas amostras são marcadas com CY3 (verde) e CY5 (vermelho), e hibridizadas às sondas no microarranjo. Apenas as moléculas que hibridizarem com avidez às sondas serão lidas como um sinal de fluorescência por um scanner, e as moléculas que não se ligarem às sondas serão retiradas após sucessivas lavagens, permitindo somente a identificação de moléculas complementares às sondas. Posteriormente a leitura no scanner de fluorescência é feita a digitalização das imagens e análise estatística.
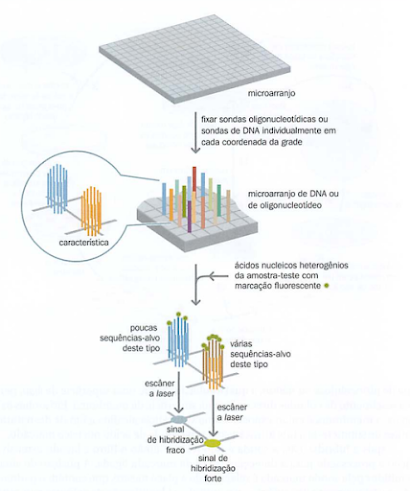
Princípio da hibridização em microarranjo:
Um microarranjo é uma superfície sólida na qual moléculas podem ser fixadas em coordenas específicas em um formato de grade de alta densidade. A hibridização em microarranjos utiliza microarranjos de DNA, ou de oligonucleotídeos, que são preparados pela fixação de milhares de sondas de fita simples sintéticas de oligonucleotídeo, ou de DNA, em posições específicas e predeterminadas na grade. Como demonstrado na figura abaixo, cada posição específica terá vários milhares de cópias idênticas de um único tipo de sonda de oligonucleotídeos ou de DNA (uma característica). Uma amostra-teste contendo um conjunto heterogêneo de fragmentos de DNA marcados, ou de transcritos de RNA, é adicionada para permitir sua hibridização com as sondas do arranjo. As sequências-alvo ligadas por algumas sondas poderão ser numerosas, resultando em um forte sinal de hibridização; para outras sondas, poderá haver poucas sequências-alvo na amostra-teste, resultando em um sinal fraco de hibridização. Após as etapas de lavagem e secagem, os sinais de hibridização para milhares de sondas diferentes no arranjo serão detectados por um scanner a laser, gerando grandes quantidades de dados a partir de um único experimento.
Subsequentemente, a hibridização em microarranjo foi utilizada para investigar a variação do DNA, bem como para traçar perfis de expressão gênica.
Quando utilizado em um ensaio de hibridização, um microarranjo consiste em milhares de sondas de DNA não marcadas ou de sondas de oligonucleotídeos fixadas a uma superfície de vidro ou de outro material adequado, em coordenadas precisamente definidas em uma grade de alta densidade. A amostra-teste, uma solução aquosa contendo uma população complexa de moléculas de DNA desnaturado ou RNA, marcadas com fluorescência, é hibridizada com as moléculas de sonda no microarranjo. Após as etapas de lavagem e secagem, a marcação fluorescente é detectada por um scanner a laser de alta resolução, e o sinal emitido a partir de cada ponto (spot) do arranjo é analisado por um software de imagem digital que converte o sinal em uma paleta de cores, de acordo com sua intensidade.
O objetivo da hibridização em microarranjo é que cada uma das milhares de sondas diferentes forme heterodúplices com sequências-alvo presentes na amostra-teste de ácidos nucleicos marcados. Em uma posição qualquer do arranjo, várias cópias idênticas de apenas um tipo de oligonucleotídeo ou de molécula de DNA estarão disponível para hibridização. Sequências-alvo estarão presentes em diferentes quantidades na amostra em estudo. A quantidade de DNA-alvo específico ligado a cada sonda dependerá da frequência daquele alvo específico na amostra-teste. Uma sonda que tem como alvo um ácido nucleico relativamente raro terá menos fluorescência ligada a ela, resultando em um fraco sinal de hibridização, enquanto posições no arranjo que são fortemente fluorescentes após a lavagem indicam sondas cujo alvo era um ácido nucleico abundante. A quantificação da fluorescência ligada em todas as posições do microarranjo gera um grande conjunto de dados que poderá refletir a frequência relativa de um grande número de sequências-alvo presentes na amostra
RESUMO:
Esboço de microarranjos de DNA
Um chip de DNA, também chamado de microarranjo de DNA, é uma ferramenta analítica que consiste em muitos fragmentos de DNA densamente colocados em uma resina ou substrato de vidro para detecção de alterações na expressão de genes em amostras. Microarranjos de DNA são usados em diversas áreas de pesquisa porque permitem análises exaustivas de várias centenas a várias centenas de milhares de genes com uma pequena quantidade de amostra e em um tempo relativamente curto. Um número crescente de artigos registrados no PubMed trata de microarranjos de DNA e chips de DNA. Muitos microarranjos de DNA são utilizados em pesquisas visando seu uso prático na prática clínica ou industrial.
Estrutura de microarranjos de DNA
Microarranjos gerais de DNA têm uma estrutura na qual muitos tipos de fragmentos de DNA conhecidos para detecção são imobilizados em um substrato com vários centímetros de lado. Várias dezenas a várias dezenas de milhares de tipos de fragmentos de ADN para detecção são imobilizados numa superfície pontual e a análise é conduzida utilizando hibridização entre estes fragmentos de DNA e DNA de genes expressos.
Uso de microarranjos de DNA
Os genes extraídos de uma amostra são rotulados como verde fluorescente, as amostras padrão de referência são rotuladas como vermelho fluorescente e, em seguida, são hibridizadas com um microarranjo de DNA. Após a hibridização, o microarranjo de DNA é lavado e lido por um scanner para detectar sinais. O exame da proporção entre sinais vermelhos e sinais verdes após a hibridização revela quais genes são expressos e seus níveis de expressão.
- Primeiro, uma sequência curta, tag (9-11 pares de bases), contém informação suficiente para identificar um transcrito único;
- Segundo, vários tags podem ser concatenados em uma única molécula e sequenciados, resultando na caracterização de muitos tags simultaneamente.
Análise seriada de expressão gênica (SAGE):
- O RNA é extraído e purificado das amostras desejadas. O RNAm é convertido em cDNA de cadeia dupla por transcriptase reversa com caudas poli-T biotiniladas como iniciadores;
- O cDNA biotinilado é fragmentado com enzimas de ancoragem que reconhecem e cortam nos seus respectivos locais de restrição que são normalmente encontrados naturalmente em cada poucas centenas de pares de bases;
- As extremidades biotiniladas do cDNA fragmentado são ligadas a esferas de estreptavidina e divididas em duas porções;
- Adaptadores apropriados com um sítio de endonuclease para marcação de enzimas são ligados a cada fragmento de cDNA na extremidade clivada;
- Enzimas de marcação são adicionadas para clivar uma pequena distância dos seus locais de endonuclease encontrados em cada adaptador, liberando o cDNA fragmentado (até 26 pares de bases) das esferas;
- As extremidades cegas de cada uma das duas porções do cDNA fragmentado são ligadas entre si, formando um ditag, e depois amplificadas por PCR;
- Após a PCR, os ditags são clivados nos seus adaptadores, concatenados e depois clonados em plasmídeos;
- Uma vez clonados, os plasmídeos são então sequenciados, mapeados de volta ao gene e posteriormente analisados quanto à expressão genética.
- Correr duas amostras de RNA(cDNA) lado a lado em um gel;
- Excisar e sequenciar bandas presentes em uma faixa, mas não na outra.
- Reduzir a complexidade das amostras produzindo o cDNA com primers que prepararão apenas um subconjunto de todos os transcritos.
Referências
DNA microarrays. In: BioNinja. Disponível em: https://ib.bioninja.com.au/options/untitled/b4-medicine/dna-microarrays.html. Acesso em: 22 nov. 2023.
Malone, Gaspar et al. PROSPECÇÃO DE GENES EM BIBLIOTECAS DE cDNA. In: Universidade Federal de Pelotas. Disponível em: https://periodicos.ufpel.edu.br/index.php/CAST/article/view/4459/3344.Acesso em: 22 nov. 2023.
Howard Y. Chang, Xin Chen. Chapter 51 - Microarray Analysis of Stem Cells and Differentiation. Editor(s): Robert Lanza, John Gearhart, Brigid Hogan, Douglas Melton, Roger Pedersen, E. Donnall Thomas, James Thomson, Ian Wilmut. Essentials of Stem Cell Biology (Second Edition), Academic Press, 2009, Pages 449-457, ISBN 9780123747297, https://doi.org/10.1016/B978-0-12-374729-7.00051-2. Disponível em: https://www.sciencedirect.com/science/article/pii/B9780123747297000512. Acesso em: 22 nov. 2023.
Capítulo 7 - Hibridação de Ácidos Nucleicos-Princípios e Aplicações. In: USP - e-disciplinas. Disponível em: https://edisciplinas.usp.br/pluginfile.php/7804304/mod_resource/content/1/Cap%C3%ADtulo%207%20-%20Hibrida%C3%A7%C3%A3o%20de%20%C3%81cidos%20Nucleicos-Princ%C3%ADpios%20e%20Aplica%C3%A7%C3%B5es.pdf#:~:text=A%20hibridiza%C3%A7%C3%A3o%20de%20%C3%A1cidos%20nucleicos%20%C3%A9%20uma%20ferramenta%20fundamental%20em,%2D%20tre%20elas%20(hibridiza%C3%A7%C3%A3o. Acesso em: 22 nov. 2023.
D. Knapen, L. Vergauwen, K. Laukens, and R. Blust, “Best practices for hybridization design in two-colour microarray analysis,” Trends Biotechnol., vol. 27, no. 7, pp. 406–414, 2009.
J. R. Pollack, C. M. Perou, a a Alizadeh, M. B. Eisen, a Pergamenschikov, C. F. Williams, S. S. Jeffrey, D. Botstein, and P. O. Brown, “Genome-wide analysis of DNA copy-number changes using cDNA microarrays,” Nat Genet, vol. 23, no. 1, pp. 41–46, 1999.
G. a Churchill, “Fundamentals of experimental design for cDNA microarrays.,” Nat. Genet., vol. 32 Suppl, no. december, pp. 490–495, 2002.
Outline of DNA microarrays. In: 3D-Gene. Disponível em: https://www.3d-gene.com/en/about/chip/.Acesso em: 22 nov. 2023.
Sarkar H, Arya S, Rai U, Majumdar SS. A Study of Differential Expression of Testicular Genes in Various Reproductive Phases of Hemidactylus flaviviridis (Wall Lizard) to Derive Their Association with Onset of Spermatogenesis and Its Relevance to Mammals. PLoS One. 2016 Mar 10;11(3):e0151150. PMID: 26963275; PMCID: PMC4795608. Disponível em: https://doi.org/10.1371/journal.pone.0151150. Acesso em: 22 nov. 2023.
Horan MP. Application of serial analysis of gene expression to the study of human genetic disease. Hum Genet. 2009 Nov;126(5):605-14. Epub 2009 Jul 10. PMID: 19590894. Disponível em: https://doi.org/10.1007/s00439-009-0719-5. Acesso em: 23 nov. 2023.























Nenhum comentário:
Postar um comentário