Olá pessoal, hoje eu trago outro tópico relevante para a biologia molecular: a organização dos genomas procarioto e eucarioto.
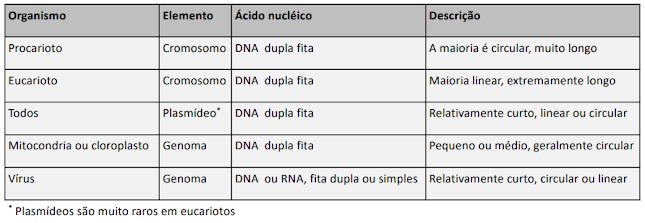
A informação genética contida no DNA de um organismo contém as instruções para todas as moléculas de RNA e proteínas que o organismo irá sintetizar, compondo o genoma do organismo. Nos procariotos, o DNA está no citoplasma e é circular - um cromossomo circular denominado nucleoide e moléculas extracromossômicas de DNA circular chamadas plasmídeos. Nos eucariotos, no núcleo das células, há o DNA associada à proteínas formando fibras de cromatina que constituem os cromossomos - embora mitocôndrias e cloroplastos também apresentem algum DNA. Vale destacar, que os vírus também apresentam material genético e ele pode ser RNA ou DNA.
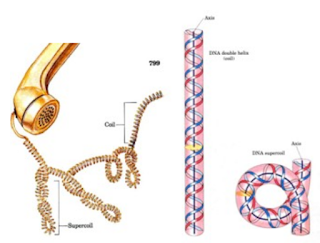
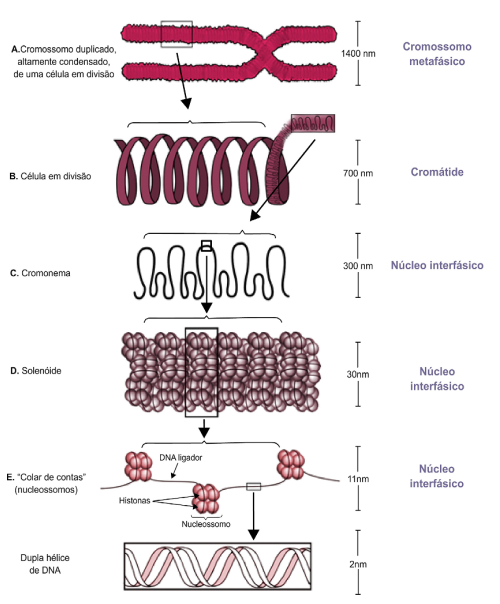
O tamanho da molécula de DNA geralmente é muito maior do que o compartimento que a contém, por isso o empacotamento do DNA no cromossomo precisa ser altamente organizado. Células eucarióticas tem muito mais DNA que células procarióticas, mas o genoma eucariótico tem alta proporção de DNA não codificador (íntrons, sequências intergênicas, DNA repetitivo).
Um gene é uma sequência de DNA que inclui todas as sequências de nucleotídeos que levam à síntese de um produto (RNAm, RNAt, RNAr etc.). É constituído por regiões codificantes e as reguladoras.
Em eucariotos, sequências codificantes podem estar interrompidas por sequências sem função codificadora.
- Éxons: codificantes; expressed sequences;
- Íntrons: não codificantes; intervening sequences; alguns organismos não apresentam íntrons, incluindo eucariotos.
O RNA é a macromolécula que pode conter a informação genética (vírus de RNA), transmitir a informação genética ou ter atividade catalítica (ribozima). O DNA transmite sua informação através do RNA, por meio da transcrição - um sistema enzimático converte a informação genética do DNA (dupla fita) para RNA (fita simples). Um RNA codificante é aquele traduzido em proteínas, quando isso não acontece ele é dito não codificante.
Operon é quando mais do que um gene é transcrito no mesmo RNA, operon é um grupo de genes sob o controle de um único operador: genes estruturais, operador, promotor e terminador. Pode haver um ou mais do que um gene sob um único promotor, mocistrônico e policistrônico, respectivamente. Bactérias podem ser mono ou policistrônicas.
Promotor é o sítio de ligação da RNA polimerase (responsável pela transcrição), terminador é a região terminadora da transcrição. Num procarioto, geralmente um promotor regula a transcrição de vários genes ao mesmo tempo, enquanto nos eucariotos, um promotor regula a transcrição de um único gene.
Organização do genoma procarioto
O DNA no organismo-modelo Escherichia coli e na maioria das bactérias apresenta as seguintes características:
- Fita dupla;
- Circular;
- Localiza-se nos nucleoides;
- Possui DNA extracromossômico, os plasmídeos, que tem origem de replicação para produzir cópias que também passam para as células-filhas na divisão celular.
Em resumo:
- Genes bacterianos são frequentemente encontrados em operons. Os genes de um operon são transcritos em grupo e possuem um promotor único;
- Cada operon contém sequências de DNA reguladoras, as quais atuam como sítios de ligação de proteínas reguladoras que promovem ou inibem a transcrição;
- Proteínas reguladoras muitas vezes ligam-se a pequenas moléculas, as quais podem tornar a proteína ativa ou inativa, alterando sua capacidade de se ligar a DNA;
- Alguns operons são induzíveis, que significa que podem ser ativados na presença de uma pequena molécula em particular. Outros são repressíveis, que significa que são ativos por padrão, mas podem ser desativados por uma pequena molécula.
- Genes de uma mesma via metabólica muitas vezes formam operons no genoma de bactérias;
- Agregam genes com funções relacionadas em operons permite que um único promotor regule a expressão de vários genes, garantindo quantidades adequadas dos produtos gênicos (proteínas);
- Como não têm núcleo, as bactérias podem executar transcrição e tradução simultaneamente, no mesmo compartimento. Isso permite aos genes em operons acoplar os processos de transcrição, tradução e formação de complexos, resultando em maior eficiência.
- Célula animal: DNA nuclear + DNA mitocondrial;
- Célula vegetal: DNA nuclear + DNA mitocondrial + DNA cloroplastidial.
- Centrômero: funciona como o local onde se prendem as proteínas que ligam os cromossomos aos microtúbulos na divisão mitótica;
- Telômero: possuem sequências localizadas nas extremidades dos cromossomos linear para dar estabilidade.
- RNA Pol I e III - muitos tRNAs, rRNAs;
- RNA Pol II - muitos genes codificadores de proteínas.
Resumo da organização genômica humana:
Cada célula humana contém muitas cópias de um genoma mitocondrial pequeno, circular, e apenas uma cópia do genoma nuclear, muito maior. Enquanto o genoma mitocondrial apresenta algumas semelhanças com o genoma compacto dos procariotos, o genoma nuclear humano é muito mais complexo em sua organização, com apenas 1,1% do genoma codificando proteínas e 95% dele correspondendo a sequências de DNA não conservadas e, geralmente, alta- mente repetitivas.
O sequenciamento do genoma humano revelou que, ao contrário do que se esperava, há relativamente poucos genes que codificam proteínas - 20.000 a 21.000, de acordo com estimativas mais recentes. Estes genes variam amplamente em tamanho e organização interna, com éxons geralmente separados por grandes íntrons, os quais frequentemente contêm sequências de DNA altamente repetitivo. A distribuição dos genes ao longo do genoma não é uniforme, com alguns genes relacionados estrutural e funcionalmente encontrados em agrupamentos, sugerindo que teriam surgido pela duplicação de genes individuais ou de segmentos maiores de DNA. Pseudogenes podem ser originados quando um gene é duplicado, e então uma das cópias acumula mutações deletérias, evitando sua expressão. Outros pseudogenes surgem quando um transcrito de RNA é reversamente transcrito e o cDNA gerado é reinserido no genoma.
A maior surpresa da era pós-genômica é o número e a variedade de RNAs não codificantes de proteínas transcritos a partir do genoma humano. Sabe-se agora que pelo menos 85% do genoma eucromático é transcrito. Às já familiares moléculas de ncRNAs (RNAs não codificantes), que atuam na síntese proteica, juntaram-se outras que desempenham funções na regulação gênica, incluindo várias classes prolíficas de pequenos RNAS regulatórios e milhares de diferentes ncRNAs longos. Nossa visão tradicional do genoma está sendo radicalmente alterada.
Regulação da expressão gênica
Regulação gênica é como a célula controla quais genes, entre os inúmeros genes presentes em seu genoma, são "ativados" (expressos). Graças à regulação gênica, cada tipo de célula possui um conjunto diferente de genes ativados - apesar do fato de que no caso de um eucarioto multicelular quase todas as células do corpo possuem exatamente o mesmo DNA.
A expressão gênica diz respeito a função decodificador de proteínas realizadas pelos genes. Para iniciar esse processo, os genes presentes no DNA são transcritos em moléculas de RNA de diversos tipos pelas enzimas RNA polimerases. A transcrição é dirigida por regiões específicas de sinalização do DNA denominadas promotores, utilizadas pelas RNA polimerazes para delimitar o início da região codificante. As etapas de transcrição são denominadas: início, elongamento e término. Existem muitas formas de regulação gênica, isto é, mecanismos para controlar quais genes serão expressos e em que níveis. No entanto, muito da regulação gênica acontece durante a transcrição.
Nos procariontes geralmente as moléculas de RNA não são processadas e os mecanismo de transcrição e tradução são praticamente simultâneos. Nos eucariontes existe o processamento de moléculas de RNA precursoras, como é o caso do RNA heterogêneo nuclear, que recebe um caput de Guanosina metilada na extremidade 5’, uma calda de poliadenina e a retirada dos íntrons e reunião do exons, realizada em um complexo sistema de processamento denominado splicing.
Eucariotos são limitados na resposta direta às variações nas condições externas, pois suas células estão organizadas em tecidos e órgãos, diferentemente de procariotos que respondem diretamente às condições ambientais. Além disso, a transcrição em eucariotos não pode ser acoplada à tradução, pois elas ocorrem compartimentos distintos. Portanto, a regulação em células eucarióticas depende do desenvolvimento e dos tecidos, estando relacionada a mecanismos como metabólitos e hormônios
A fosforilação pode regular os fatores de transcrição, que são proteínas que se ligam ao DNA e propiciam a capacidade de controlar a expressão gênica sob diferentes estímulos metabólicos ou condições de crescimento.
Quando os genes são expressos?
- ''Housekeeping'' (proteínas constitutivas: produzidas em velocidade e quantidade constantes, independente do estado metabólico do organismo);
- Desenvolvimento (só em um período da vida);
- Tipo específico de célula;
- Resposta a alterações (hormônios, vitaminas, variação de temperatura etc.). Proteínas induzíveis: aumentam/diminuem a quantidade em resposta a sinais recebidos pelo meio.
- Proteoma: conjunto completo de proteínas que podem ser expressas pelo material genético;
- Transcriptoma: coleção completa de sequências transcritas do genoma, compreendendo os RNAsm e ncRNAs (RNAs não codificantes, do inglês “non coding RNA”);
- Genoma: conjunto de material genético em um conjunto cromossômico.
Regulação da expressão gênica em procariotos
Recordando as características dos genomas procarióticos:
- Ausência do complemento diploide de genes;
- Uso de quase todo o genoma na codificação e na regulação praticamente sem a presença de DNA redundante;
- O tamanho do gene corresponde ao tamanho da proteína (ausência de íntrons);
- Tendência de apresentar genes codificando funções relacionadas agrupados em unidades transcrionais (operons);
- Ocorrência, com frequência aproximadamente igual, de sequências codificantes em ambas fitas de DNA;
- Unidades genéticas acessórias (plasmídeos, bacteriófagos, elementos de transposição).
- Possui ~4600 genes;
- Alguns codificam proteínas sempre necessárias (exemplos: enzimas envolvendo glicólise, tRNA, rRNA, proteínas ribossomais, RNA polimerase etc.);
- Genes constantemente transcritos são chamados de genes constitutivos;
- Outros genes são transcritos somente quando a célula necessita dessas proteínas, esses são os genes induzíveis.
- Seu hábito alimentar determina completamente os nutrientes disponíveis para essa bactéria;
- Supre sua inabilidade de alterar o ambiente externo sendo internamente flexível;
- Se a glicose estiver presenta, ela a utiliza para gerar energia (ATP);
- Se não há glicose, ela utiliza lactose, arabinose. maltose ou qualquer outro açúcar;
- Ela responde às mudanças ambientais alterando sua bioquímica, isso ter um alto preço pelo consumo alto de energia;
- Essa bactéria mantém sua flexibilidade bioquímica enquanto otimiza a eficiência de energia através da regulação gênica;
- Se uma fonte de carbono aparece no ambiente, genes codificando proteínas que metabolizam oi carbono são rapidamente transcritos e traduzidos;
- Quando a fonte de carbono desaparece, os genes que codificam essas proteínas são desligados.
- Operon lac (lactose): sistema induzível;
- Operon ara (arabinose): sistema induzível;
- Operon trp (triptofano): sistema repressível.
- Assim, no metabolismo da lactose por E. coli a primeira enzima produzida pelo operon é a beta-galactosidade, uma enzima capaz de clivar a lactose em glicose e galactose, que assim servirão como fonte de carbono para a célula.
- A beta-galactosidase é uma enzima indutível, ou seja, sua expressão varia de acordo com as necessidades celulares e, caso a bactéria esteja crescendo em meio rico em lactose, sua expressão será alta; no entanto, caso a fonte de carbono seja outro carboidrato, sua expressão será reduzida. A outra enzima do operon é a permease, que, como seu próprio nome indica, é a enzima responsável pelo transporte de lactose do meio extracelular para o meio intracelular através da membrana da célula bacteriana, pois a lactose, como a maioria dos carboidratos, não é capaz de atravessar a bicamada lipídica sem uma proteína carreadora.
- A última enzima, a tiogalactosídeo transacetilase tem função de inativar galactosídeos tóxicos, que também acabam sendo transportados para dentro da célula. A expressão dessas três proteínas é regulada por um outro gene (gene I) que codifica para um regulador Lac. O gene I é capaz de codificar um repressor que está faltando ou está inativado nos organismos com o gene I-. A região promotora (P), como visto anteriormente, é o local onde a RNA Polimerase se liga para começar a transcrição. Num operon, além da região promotora existe também o operador (O), os quais formam os locais de controle do operon. Na presença do produto do gene I, o operon é incapaz de ser codificado, pois o repressor está ligado ao operador , no entanto, caso o indutor, a própria lactose esteja presente, essa se liga ao repressor o qual não se liga ao DNA, não bloqueando a síntese das proteínas envolvidas no metabolismo da lactose.
- Gene I - codifica proteína repressora;
- Sítio P - sítio no DNA ao qual se liga a RNA polimerase para início da transcrição;
- Sítio O - sítio do DNA ao qual se liga o repressor;
- Gene Z - beta-galactosidase (lactose = glicose + galactose);
- Gene Y - permease (transporta lactose para a célula);
- Gene A - transacetilase (transfere o grupo acetil de acetilCoA para a beta-galactosidase).
Regulação da expressão gênica em eucariotos
Diferentes células em um organismo multicelular podem expressar conjuntos de genes muito diferentes, apesar de possuírem o mesmo DNA. O conjunto de genes expressos em uma célula determina o grupo de proteínas e RNAs funcionais que ela possui, conferindo-lhe suas características únicas.
Em eucariontes, como os humanos, a expressão gênica envolve várias etapas e a regulação de genes pode acontecer em qualquer uma delas. Contudo, muitos genes são regulados primariamente no momento da transcrição.
O mecanismo de regulação em eucariotos está basicamente sob um controle positivo. Isso porque, se os genes estivessem sob um controle negativo, haveria necessidade da síntese de aproximadamente 100 mil repressores para se ligarem aos operadores gênicos e impedirem a transcrição das cerca de 100 mil proteínas, que seria o número estimado de proteínas anteriormente à conclusão do Projeto Genoma Humano. Considerando um controle positivo, as proteínas regulatórias podem se ligar aos sítios reguladores, estimular a transcrição e se desligar ligando-se em outro local.
Aqui é a observação de que existem nesse mecanismo regulatório os elementos de atuação em “cis” e os elementos regulatórios de ação em “trans”:
- Os elementos regulatórios em “cis” são aqueles que desempenham funções regulatórias da expressão gênica no mesmo cromossomo da sequência de um gene que irá ser transcrito, como as regiões promotoras dos genes, seus elementos proximais e os distais, destacando-se os reforçadores ou enhancers. Os silenciadores também são sequências em “cis” localizadas na molécula de DNA, cuja função é reduzir ou reprimir as taxas de transcrição de um gene;
- Os elementos em “trans” são aqueles que desempenham funções regulatórias à distância da sequência pela qual foram codificados, atuando normalmente em cromossomos diferentes dos quais foram produzidos. Os transativadores correspondem às proteínas produzidas por RNA expressos de cromossomos diferentes daqueles em que irão regular, por isso a denominação “trans”. Dentro dessa classe estão as proteínas que atuam como fatores de transcrição (TFs), que são capazes de recrutar outras proteínas, como os coativadores, visto que a afinidade da RNA polimerase ao promotor é baixa, havendo a necessidade de ligação das proteínas regulatórias para aumentar a afinidade da RNA polimerase ao promotor e iniciar a transcrição. Para regular os níveis de transcrição em eucariotos, o modelo é complexo envolvendo a regulação gênica por cascata proteica dada pelos fatores gerais de transcrição (GTFs).
Os hormônios e seus fatores de transcrição representam uma classe de reguladores da expressão gênica em “trans” de ligação a sequências denominadas elementos de resposta a hormônio (HRE) (cis), que correspondem aos sítios de ligação no DNA dos receptores proteicos de ligação a hormônios. As proteínas receptoras de hormônio se localizam no citoplasma, mas atuam no núcleo da célula, possuindo domínio de ligação a ligante e domínio de ligação ao DNA, levando à transcrição de genes de resposta a hormônios.
Dentre os exemplos dessa classe estão as proteínas receptoras de andrógeno (AR). Essa proteína pertence à família dos receptores nucleares e possui dois domínios dedos de zinco de ligação ao DNA. No citoplasma está na sua forma inativa associada às chaperonas que impedem a sua degradação. Quando a testosterona entra na célula, no citoplasma é convertida em DHT (dihidrotestosterona) pela ação da enzima 5-alfa-redutase, e a DHT se liga ao domínio de ligação a ligante do receptor de andrógeno. O AR complexado à DHT passa para o núcleo e na forma de homodímero levam à transcrição dos genes regulados por hormônio pela ligação ao ARE (elemento de resposta a andrógeno) .
Em resumo:
Em eucariotos, genes que codificam proteínas possuem éxons (sequências traduzidas) e íntrons (removidos do RNAm antes da tradução - splicing). O sítio de início da transcrição pode ser um promotor principal (perto da TATA box) ou um promotor secundário (distante da TATA box).
Também são importantes os reguladores, que se ligam a essas sequências e dão início a uma série de reações que, por último, especificam quais genes serão transcritos e em que taxa - acentuadores (enhancers), silenciadores, isoladores e os fatores de transcrição. Os elementos regulatórios de ação cis são os acentuadores, silenciadores e isoladores, enquanto os elementos regulatórios de ação trans são os fatores de transcrição.
Apontamentos:
- Ativadores são reguladores positivos, que aumentam a transcrição de um gene. Repressores são reguladores negativos, que reduzem ou anulam a transcrição de um gene;
- Os ativadores e os repressores que ligam-se a sequências na mesma fita de DNA do promotor são elementos cis;
- Os ativadores e os repressores que são proteínas sintetizadas por genes localizados em outras regiões do genoma são elementos trans;
- A ativação da transcrição ocorre por proteínas ligadas a sequências próximas ou distantes do promotor;
- Ligação do repressor a um sítio que se sobrepõe à região de ligação da polimerase;
- O ativador recruta a polimerase;
- Enhancer é a região de ligação do estimulador de transcrição;
- Os ativadores e os repressores de organismos superiores possuem regiões (domínios) diferentes para ligação ao DNA e para ativação ou repressão;
- Algumas vezes, a função de ligação ao DNA e de ativação são desempenhadas por moléculas diferentes;
- Fatores de transcrição basais são moléculas reguladoras trans e fatores regulatórios cis são os acentuadores e repressores da transcrição.
Referências
ALBERTS B, BRAY D, JOHNSON A et al. Molecular Biology of the cell. New York: Garland Publishing, Inc., 2014. 6th. ed., 1268p.STRACHAN, Tom; READ, Andrew P. Genética Molecular Humana. 2aed., Porto Alegre (RS): ARTMED, 2002. XXIII, 576p. ISBN 857307907X (enc.)
SILVA, A. E. Organização do Genoma Humano. In: SlidePlayer. Disponível em: <https://slideplayer.com.br/slide/3446959/>. Acesso em: 25 set. 2023.
Genética de procariotos. In: USP: e-disciplinas. Disponível em: <https://edisciplinas.usp.br/pluginfile.php/7669441/mod_resource/content/1/genetica.pdf>. Acesso em: 25 set. 2023.Organização dos genomas dos procariotos e dos eucariotos. In: SlidePlayer. Disponível em: <https://slideplayer.com.br/slide/1575555/>. Acesso em: 25 set. 2023.
Conceitos de Biologia Molecular. In: IQ-USP. Disponível em: <https://www.iq.usp.br/setubal/qbq102/2022/aula8transcricao.pdf>. Acesso em: 25 set. 2023.
Replicação do DNA e transcrição. In: CESAD - UFS. Disponível em: <https://cesad.ufs.br/ORBI/public/uploadCatalago/15352716022012Genetica_Basica_Aula_6.pdf>. Acesso em: 25 set. 2023.
Regulação gênica em bactérias. In: Khan Academy. Disponível em: <https://pt.khanacademy.org/science/ap-biology/gene-expression-and-regulation/regulation-of-gene-expression-and-cell-specialization/a/overview-gene-regulation-in-bacteria>. Acesso em: 25 set. 2023.




















Nenhum comentário:
Postar um comentário